近日,学校高性能计算云平台针对AlphaFold2、RoseTTAFold等软件开展了蛋白质结构预测计算环境建设工作,取得阶段性成果,基本建成AlphaFold2批量结构预测平台。
校级公共技术平台一直积极推进和探索基于大型仪器设备平台的学科交叉。高性能计算云平台作为校级公共技术平台的重要组成部分,采用云计算行业中先进的容器技术,实现了计算环境的快速配置和构建,并同微生物技术研究院深度合作,基于生物信息大数据和深度学习网络的蛋白质结构预测技术,建设了AlphaFold2批量结构预测平台。平台充分发挥了在设备资源和软件系统构建技术方面的优势,将整个AlphaFold 2应用模块、模型数据和应用软件集成在统一的虚拟环境中,建设了2.2TB配套的数据库资源,并利用平台配置的NVIDIA V00为用户提供优秀的并行计算算力支持。用户只需要简单的虚拟环境导入即可使用,省去了数据下载、软件模块安装配置等诸多繁琐程序,同时也极大缓解了校园网国际出口带宽和流量压力。
相较于国内其他高校的高性能计算平台,山东大学高性能计算云平台借助平台的资源共享理念和技术支持措施,提供了更加友好和丰富的用户交互模式。全校所有用户,均可登录高性能计算云平台,使用AlphaFold 2的全部功能组件,实现了一次构建、多次部署、全校共享、使用简便的良好效果。
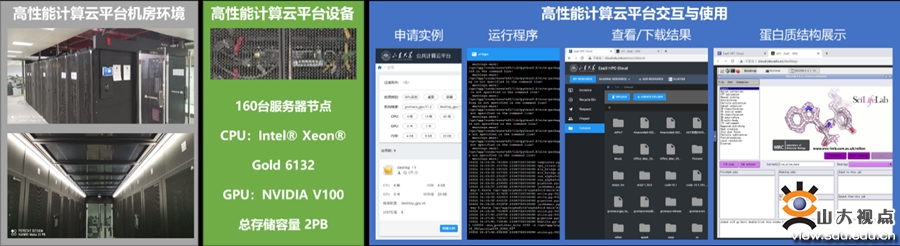
高性能计算云平台硬件与简洁的用户使用界面
在AlphaFold2软件测试的过程中,选择了来自12个不同家族的蛋白质作为批量测试目标。根据已知结构,测试样本中含不同二级和超二级构象,序列长度分布在200~500个氨基酸,总耗时约10小时。将预测结构与实验测得的蛋白质结构进行结构比对后,得到RMSD(Root Mean Squared Error)的平均值为0.290。
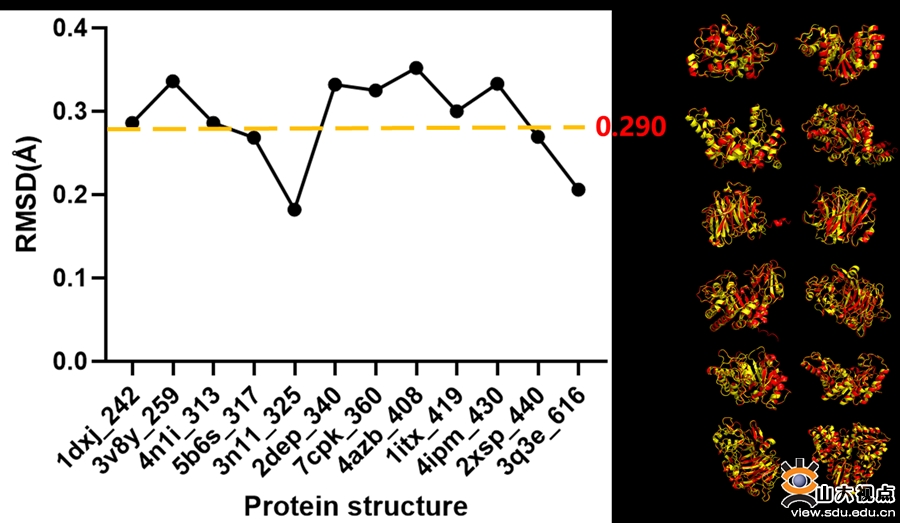
结构预测准确性
AlphaFold2批量结构预测平台的建成将使山东大学蛋白质结构测定分析能力更加完整,是X射线晶体衍射(X-ray crystallography)平台、核磁共振波谱(NMR spectroscopy)分析平台及电子显微镜技术(electron microscopy)平台之后的,第四种蛋白质结构解析平台与技术,将大大推动生物大分子结构与功能的研究向更通用、更高效、更深入。